技术摘要:
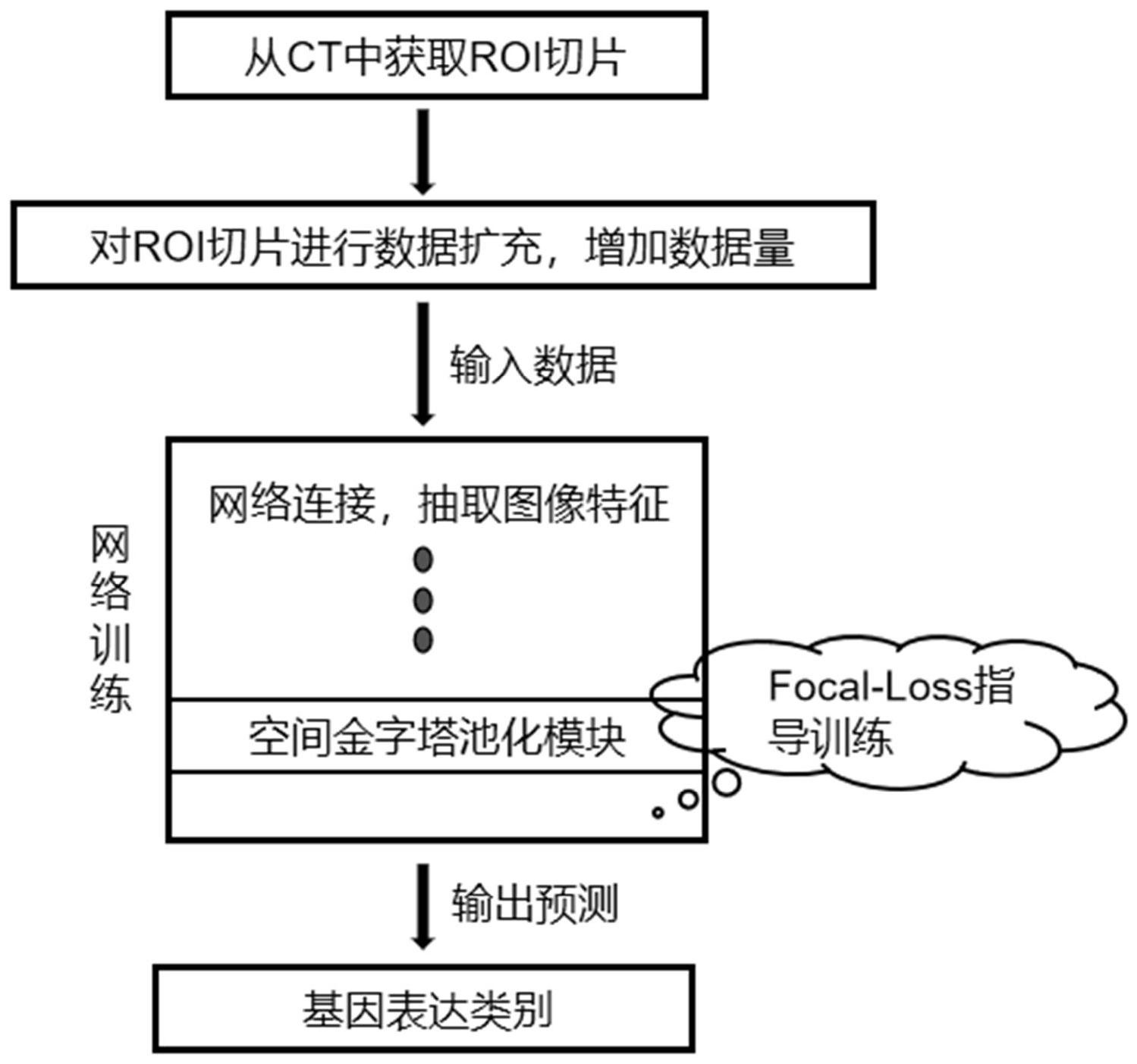
本发明公开了一种基于癌症CT图像自动预测基因表达类别的方法,该方法包括以下步骤:a)获取ROI切片并将数量扩充48倍;b)基于DenseNet‑12和空间金字塔模块构建神经网络;c)使用聚焦损失函数进行训练;d)对模型预测综合评判得到最终的预测结果。本发明采用的数据扩充 全部
背景技术:
国内外最近的研究显示,癌症CT图像提取的特征与某些基因表达模式相关。如 Shinagare等人在2015年验证得到肿瘤边缘、结节增强和肿瘤内血管与VHL突变之间的关 联,Karlo等人在2014年提出PBRM1和SETD2两类基因突变主要见于实体(非囊性)肾透明细 胞癌病例中。近两年来,越来越多的人开始在这上面进行探索。如2018年Mohammad等人利用 多示例学习的CNN网络来检测肾透明细胞癌中的4中最常见基因突变的检测;2019年国内某 高校利用3D神经网络预测肺癌中的EGFR突变,并取得了超过传统影像组学的效果;Nicolas Coudray等人利用神经网络对非小细胞肺癌中的多个基因(STK11、EGFR、SETBP1、TP53、 FAT1、KRAS、KEAP1、LRP1B、FAT4、NF1)进行预测,并讨论和分析实验结果,证明了神经网络技 术能在更多的肿瘤类型与基因型上探索的可行性。 但是现有的这些方法都使用了大量的医学数据,而在实际情况中往往难以搜集到 具有基因突变状态金标准的特定肿瘤CT数据集。并且,由于肿瘤大小、位置、形状的不同,现 有的方法都会将其重采样为固定的尺寸进行训练,这无疑会损失图像的精度以及忽略了肿 瘤个体间的差异。此外,CT序列的肿瘤边缘(即轴向的首端和末端)一般包含较少的肿瘤部 分,这些切片层面很难进行特征的识别,现有的方法对此也都没有进行关注。
技术实现要素:
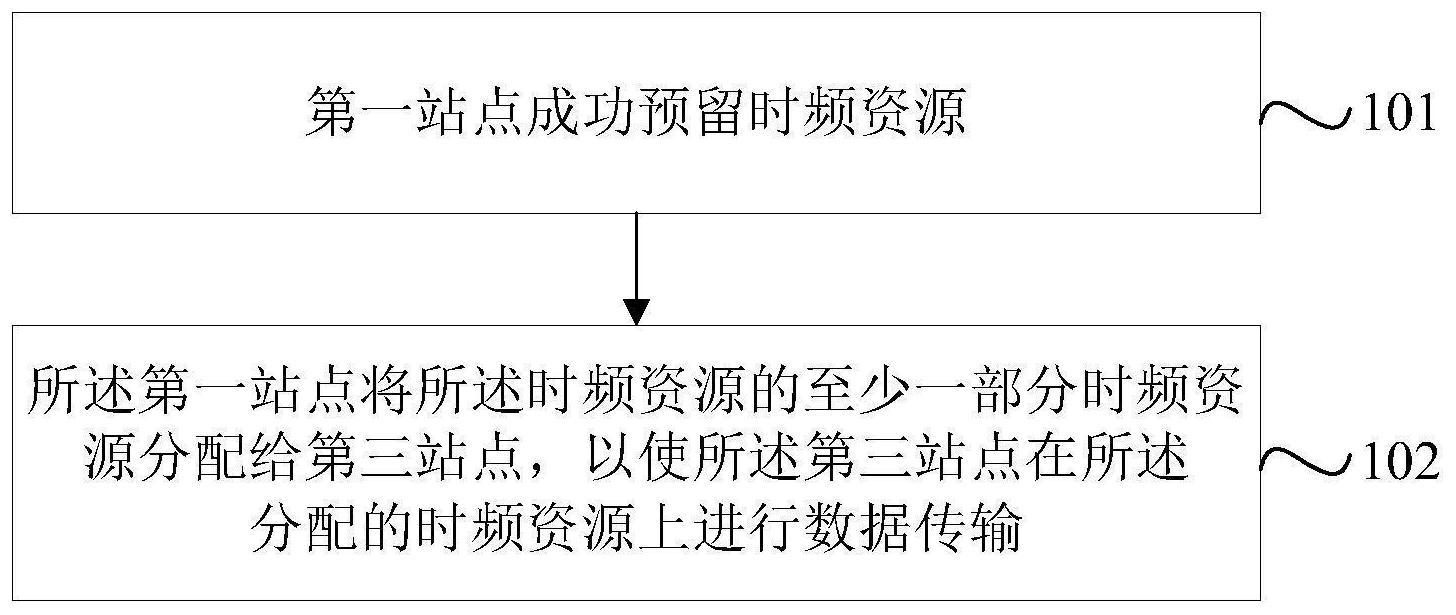
本发明的目的是针对现有技术的不足而提出的一种基于癌症CT图像自动检测基 因表达类型的辅助诊断方法。该方法一方面使少量的数据扩充后发挥更大的作用,另外采 用金字塔模块取消输入尺寸必须固定的限制,以及使用Focal-loss函数关注那些难以预测 的切片,最终训练得到准确和高效的预测结果。 实现本发明目的的具体技术方案是: 一种基于癌症CT图像自动预测基因表达类别的方法,该方法包括以下具体步骤: 步骤1:获取ROI切片并将图像数量扩充48倍; 步骤2:基于DenseNet-12和空间金字塔模块构建神经网络; 步骤3:将步骤1扩充的图像作为输入,使用步骤2构建的神经网络进行训练,训练 的损失函数采用聚焦损失即Focal-Loss; 步骤4:使用步骤3训练后的网络模型进行预测,得到每一份输入图像的基因表达 类别预测结果,即过表达、不表达或者阳性、阴性,并汇总同属一个CT序列的所有输入图像 的预测结果,得到该CT序列整体的预测结果。 所述步骤1具体包括: 4 CN 111583271 A 说 明 书 2/6 页 步骤A1:将完整的CT序列,抽取出包含肿瘤的切片,并根据肿瘤在切片上的位置以 及大小,裁剪得到一个感兴趣区域即ROI立方体,ROI立方体为包含完整肿瘤的切片序列; 步骤A2:对裁剪得到ROI切片序列,其大小为n×w×h,n为序列层数,w为宽度,h为 高度,将相邻的3张切片堆叠形成一组具有3个通道的数据,其大小为3×w×h;并将每组3通 道数据内的3张ROI切片打乱堆叠的顺序,形成6种堆叠形式,将得到的数据记为A,其大小为 n'×3×w×h,其中n'=6*n; 步骤A3:对步骤A2的数据A进行转置,得到转置后的数据B,其大小为n'×3×h×w; 步骤A4:对步骤A2的数据A进行上下翻转,形成数据C,大小为n'×3×w×h; 步骤A5:对步骤A2的数据A进行左右翻转,形成数据D,大小为n'×3×w×h; 步骤A6:对步骤A2的数据A进行1次90°旋转,形成数据E,大小为n'×3×h×w; 步骤A7:对步骤A2的数据A进行2次90°旋转,形成数据F,大小为n'×3×w×h; 步骤A8:对步骤A3的数据B进行1次90°旋转,形成数据G,大小为n'×3×w×h; 步骤A9:对步骤A3的数据B进行左右翻转,形成数据H,大小为n'×3×h×w,至此 ROI切片序列在不改变本身图像性质的同时,图像的数量扩充为原来的48倍,即为A B C D E F G H的和。 所述步骤2具体包括: 步骤B1:将DenseNet-12第一层卷积层的卷积核调整为5*5,步长调整为1; 步骤B2:移除DenseNet-12的第一层池化层,并将步骤B1的卷积层直接连接第一个 Dense Block; 步骤B3:第一个Dense Block包含6层Dense Layer,每一层Dense Layer由顺序连 接的卷积层(Conv)、批标准化层(BatchNorm)和激活层(ReLU)组合形成,将其中所有卷积层 的卷积核调整为3*3,步长调整为1; 步骤B4:将第一个Dense Block之后的transition层调整为2*2的最大池化; 步骤B5:在transition层后连接第二个Dense Block,设置与步骤B3中的Dense Block相同; 步骤B6:第二个Dense Block之后连接具有4个池化核的空间金字塔池化模块,即 SPP:Spatial Pyramid Pooling;SPP用于提取多层次的图像特征,输出1*1、2*2、3*3、4*4共 4种大小的特征映射; 步骤B7:SPP后顺序连接3层全连接层,相邻全连接层中间设置丢参率为0.5的 Dropout层,保证逐步的筛选出对于基因表达类型预测关联性最大的特征;其中第一层全连 接层的输入单元数量为4200,输出单元数量为4200;第二层全连接层的输入单元数量为 4200,输出单元数量为1000;第三层全连接层的输入单元数量为1000,输出单元数量为2。 所述步骤3具体包括: 步骤C1:步骤1得到的数据A B C D E F G H记为data-1,将data-1经过中心裁剪 得到切片大小为64*64的数据,记为data-2; 步骤C2:将data-2送入步骤2的网络中使用随机梯度下降法进行50轮训练,训练设 置batch为64,训练所需的损失函数使用聚焦损失即Focal-Loss,其计算公式如下: 5 CN 111583271 A 说 明 书 3/6 页 其中y是真实数据的基因表达类别标签,为1或者0,其中y=1表示该数据的基因表 达类别为“过表达”或者“阳性”;y=0表示该数据的基因表达类别为“不表达”或者“阴性”; 公式中y'是模型对于每一份输入图像正确预测的概率值,为0到1之间的一个小 数;其中y'越接近1,表示模型对输入图像正确预测的可能性越高; 由于实际情况中基因表达类别为“过表达”的概率要大于“不表达”的概率,即在训 练数据的分布中,y=1的数据数量要小于y=0的数据数量,这就导致训练过程中两种类别 的数据量不均衡;不同类别的数据量不均衡将导致网络并很难从数据中学习规律;α为可调 整的参数,参数值的范围在0到1之间,用以解决数据量不均衡问题;具体来说,当α设置为大 于0.5且小于1时,1-α就相应的为大于0且小于0.5,那么在上述公式中y=1的数据就会产生 更大的影响,y=0的数据就会产生更小的影响,从而使网络对于“过表达”类别的数据更多 的关注; 另外,在肿瘤切片轴向的首端或者末端往往难以挖掘有效的图像特征,因为首端 和末端的图像都包含的是肿瘤的边缘区域,仅仅带有少量的肿瘤组织信息,因此这部分图 像在模型中很容易预测错误。而公式中的参数γ用于解决这个问题。具体将γ设置为2,那 么就会使模型预测产生的损失进行平方,从而对预测错误的图像产生更大的损失,指导网 络在训练过程中给予这些图像更多的注意力,使模型的特征学习能力更强大。 该步骤经过50轮的训练获得模型M1; 步骤C3:将data-1经过中心裁剪得到切片大小为100*100的数据,记为data-3;并 将data-3送入步骤C2得到的模型M1中训练,训练设置同步骤C2,50轮训练后获得模型M2; 步骤C4:将data-1送入步骤C3获得的模型M2中训练,获取准确率最高的最终模型 M3;训练设置batch为1,同样采用Focal-loss的随机梯度下降法进行训练。 所述步骤4具体包括: 步骤D1:将CT序列进行ROI立方体提取,并将ROI切片序列相邻3张切片组合形成3 通道的输入数据,记为Input; 步骤D2:将Input输入训练好的模型M3,预测得到每一份3通道数据的基因表达类 别,所述类别为过表达、不表达或者阳性、阴性; 步骤D3:设置阈值为0.5,对于Input中的所有3通道数据的基因表达类别预测进行 综合评判,类别预测为50%以上的即为最终的预测结果。 本发明的有益效果在于: 本发明具有易行性,只需要人工对CT图像提取肿瘤区域即可,不需要固定的大小 尺寸,获取的任意大小切片均可进行训练。本发明具有非侵入性,传统的基因型鉴定需要活 检和序列检测,这是侵入性的,可能会受到难以获得组织样本、以及患者风险加大的影响。 在此,提出了的深度学习方法,通过非侵入性计算机断层扫描(CT)预测肿瘤中基因的表达 状态。本发明具有高效性,一方面使少量的数据扩充后能发挥更大的作用,另外考虑肿瘤个 体间的差异,所以采用4个维度金字塔模块取消输入尺寸必须固定的限制,同时还能把握到 多层次的图像特征。最后使用Focal-loss函数关注肿瘤边缘那些难以识别特征的切片,通 6 CN 111583271 A 说 明 书 4/6 页 过3个尺寸不同batch的训练方式得到准确和高效的预测结果。 附图说明 图1为本发明的流程图; 图2为本发明的ROI切片获取示意图; 图3为本发明的数据扩充示意图; 图4为本发明的网络框架示意图。