技术摘要:
本申请涉及一种针对细胞分选后的单细胞全基因组扩增方法,包括步骤:(a)对含有目标微量细胞的样本进行细胞分选,获得目标微量细胞;(b)将目标微量细胞采用温和裂解体系进行裂解,获得DNA,所述温和裂解体系包含温和裂解配方、较低的裂解温度和较长的裂解时间;(c)对步 全部
背景技术:
研究发现人体内有一些含量极低但具有重要研究价值的细胞,如:孕妇外周血中 含有极少量的胎儿有核红细胞,对该细胞的基因分析可用于胎儿无创产前检测;血液中的 循环肿瘤细胞,该细胞可用于肿瘤早期检测及辅助治疗;等等。由于这些细胞含量极低,因 此对这些微量细胞的扩增是重要环节,目前采用的技术为单细胞全基因组扩增技术,该技 术的理想是将单细胞/微量细胞中的DNA完整、准确的扩增很多倍、用于后续的基因分析。单 细胞全基因组扩增技术目前主要有三种方法:1 .MDA (Multiple displacement amplification,多重置换扩增),它是2001年由Laskin团队发明,具有很强的链置换性、可 在等温条件下扩增;2.MALBAC(multiple annealing and looping-based amplification cycles,多次退火环状循环扩增技术),它是2012年由谢晓亮团队发明,拟线性的扩增过程 降低了指数扩增的序列偏好性,MALBAC的起始模板量可低至皮克级,并且具有很好的扩增 均一性及较小的扩增偏差;3.ChromInst扩增技术,它是2016年由亿康基因在MALBAC的基础 上进行优化得到的一个试剂盒产品,ChromInst为该试剂盒的商标,业界简称ChromInst扩 增技术,ChromInst扩增的起始模板量也可低至皮克级,并且操作步骤简化,能够实现一步 成库,并且具有良好的扩增效果、基因组覆盖率及较小的扩增偏差。 但是,由于对所述单细胞/微量细胞的分离、捕获过程应用的细胞分选技术,会对 后续单细胞全基因组扩增造成干扰,导致扩增效率低、偏好性显著、扩增错误率高的问题, 影响基因分析结果的准确性。具体的,细胞分选是将一种细胞从多种细胞中分离出来的技 术,常见的是利用荧光标记的特性来分离细胞,例如荧光激活细胞分选技术(FACS, fluorescence-activated cell sorting),细胞分选时使用的荧光标记物质、或固定剂如: 碘化丙啶、多聚甲醛,会造成细胞损伤、核酸片段化、DNA-蛋白质交联,从而导致扩增效率低 (扩增产物少,总量不足1μg)、具有显著的偏好性;另外,固定剂的使用,还会造成脱氨基作 用导致的C碱基变成U碱基,从而导致扩增错误率高,这也是单细胞/微量细胞捕获和扩增 后,对下游应用的主要限制因素。 因此,本领域迫切需要一种针对细胞分选后的单细胞全基因组扩增方法,能够降 低细胞分选对后续单细胞全基因组扩增的影响,从而使扩增效率更高、扩增均匀性更好、扩 增错误率低。
技术实现要素:
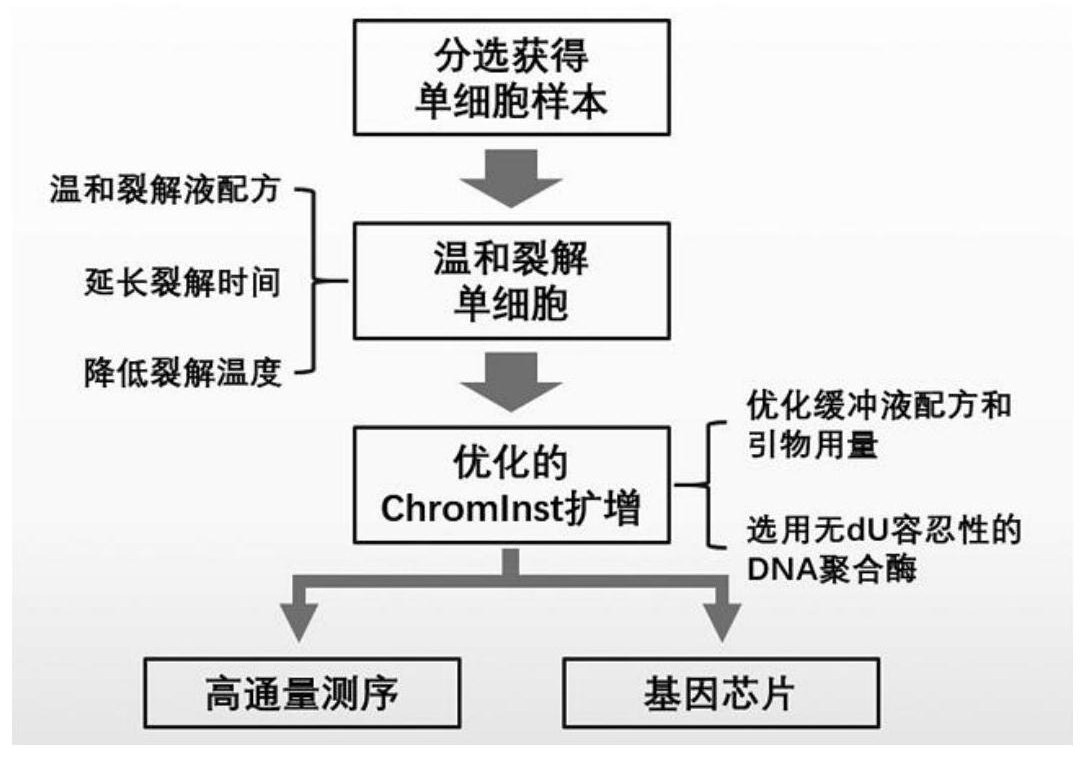
本发明的目的在于,提供一种针对细胞分选后的单细胞全基因组扩增方法,能够 降低细胞分选后的细胞损伤、核酸片段化、DNA-蛋白质交联以及碱基变异,从而提高扩增效 率、扩增均匀性以及降低扩增错误率。 3 CN 111575346 A 说 明 书 2/10 页 本申请人经过长期广泛深入的研究,通过大量筛选和测试,首次发现,对于孕妇外 周血中的胎儿有核红细胞、血液中的循环肿瘤细胞、卵细胞、生殖细胞、肿瘤组织细胞等人 体内微量细胞,进行细胞分选后,通过优化的裂解体系和扩增体系,具体的,通过利用温和 的裂解体系及优化的扩增体系,进行裂解及单细胞全基因组扩增,可获得更高的扩增效率、 更好的扩增均匀性、且扩增错误率低。在一些实施方案中,可实现在单细胞水平有效扩增2- 4ug的全基因组产物、覆盖度>90%、重复单一位点扩增成功率>95%。 具体地,对含有目标微量细胞的样本进行细胞分选后,采用温和裂解体系对目标 微量细胞进行裂解,温和裂解体系采用温和裂解配方,可以减少细胞损伤,同时降低裂解温 度并且延长裂解时间,可以减少DNA-蛋白质交联;然后将获取的DNA采用优化的扩增体系进 行扩增,优化的扩增体系是在ChromInst技术的基础上进行优化,在本申请中称为优化的 Chromlnst体系,所述优化的Chromlnst体系包含优化的扩增缓冲液和引物浓度,使之更适 合核酸短片段的扩增并且有针对性的提高短片段的扩增效率,可以降低核酸片段化及核酸 片段化的影响,从而提高扩增效率及扩增均匀性;更重要的,优化的Chromlnst体系应用了 无dU容忍性的DNA聚合酶,该DNA聚合酶对于dU碱基不耐受,不能识别DNA模板上的U碱基,因 此受细胞分选固定剂作用C-U的错误突变在扩增阶段就会被排除,从而极大的降低了扩增 错误率;扩增后的产物可用于高通量测序平台测序、芯片平台测序及数据分析。在此基础 上,本发明人完成了本发明。 因此,本申请的一方面,提供一种针对细胞分选后的单细胞全基因组扩增方法,所 述方法包括步骤: (a)对含有目标微量细胞的样本进行细胞分选,获得目标微量细胞,其中所述样本 为来源于人类或哺乳动物,所述目标微量细胞为人体内含量极低但具有重要研究价值的细 胞,例如孕妇外周血中的胎儿有核红细胞、循环肿瘤细胞、卵细胞、生殖细胞、肿瘤组织细 胞;其中所述细胞分选为常规的荧光标记细胞分选技术,例如荧光激活细胞分选技术 (FACS,fluorescence-activated cell sorting)、荧光流式细胞分选技术。 (b)将目标微量细胞采用温和裂解体系进行裂解,获得DNA,其中所述温和裂解体 系包含温和裂解配方,减少细胞损伤;同时降低裂解温度并且延长裂解时间,减少DNA-蛋白 质交联。 在一些实施方案中,所述温和的裂解配方进一步包括:0.4%-0 .6%的NP-40、 0.2%-0.3%的脱氧胆酸盐、和低浓度的蛋白酶K,进一步的,所述蛋白酶K的浓度由常规的 100μg/mL降低至15-25μg/mL。 在一些实施方案中,所述温和裂解体系的裂解温度较低,进一步的,所述裂解温度 由常规的55℃降低至3-6℃;所述温和裂解体系的裂解时间较长,进一步的,所述裂解时间 由常规的1小时延长至2.5-3.5小时。 (c)对步骤(b)获取的DNA采用优化的扩增体系进行扩增,获得扩增产物,其中所述 优化的扩增体系为优化的Chromlnst体系,所述优化的ChromInst体系包含优化的扩增缓冲 液和引物浓度、以及无dU容忍性的DNA聚合酶。 在一些实施方案中,所述优化的扩增缓冲液和引物浓度,更适合核酸短片段的扩 增并且能够有针对性的提高短片段的扩增效率,降低了核酸片段化及核酸片段化的影响; 所述无dU容忍性的DNA聚合酶,该DNA聚合酶对于dU碱基不耐受,不能识别DNA模板上的U碱 4 CN 111575346 A 说 明 书 3/10 页 基,因此受细胞分选的固定剂作用C-U的错误突变在扩增阶段就会被排除,极大的降低了扩 增错误率。 在一些实施方案中,所述优化的扩增缓冲液进一步包括:45-55mM的Tris-HCl (pH7.4)、23-27mM的KCl、18-22mM的MgCl2、3-7mM的(NH4)2SO4、0.8-1.2mM的dNTPs、22-27μM 的Pre-primer,所述优化的引物浓度为:9-18ng/ul的泓迅181114引物。 在一些实施方案中,所述无dU容忍性的DNA聚合酶为:0.05-0.1U/uL的Deep Vent EXO- R DNA polymerase,可以有效的避免由于脱氨基作用导致的G/C到T/A的假阳性突变。 在一些实施方案中,扩增产物可用于高通量测序平台测序、芯片平台测序及数据 分析。 本申请的另一方面,提供一种针对细胞分选后的单细胞全基因组扩增的试剂盒, 所述试剂盒包括:温和裂解配方、优化的ChromInst体系。 在一些实施方案中,所述温和裂解配方进一步包括:0.4%-0.6%的NP-40、0.2%- 0.3%的脱氧胆酸盐、和低浓度的蛋白酶K,进一步的,蛋白酶K的浓度由常规的100μg/mL降 低至15-25μg/mL;目标微量细胞进行裂解时,与温和裂解配方相配合的,需要使用较低的裂 解温度和较长的裂解时间,所述裂解温度由常规的55℃降低至3-6℃,所述裂解时间由常规 的1小时延长至2.5-3.5小时。 在一些实施方案中,所述优化的ChromInst体系包含优化的扩增缓冲液和引物浓 度、以及无dU容忍性的DNA聚合酶,所述优化的扩增缓冲液和引物浓度,更适合核酸短片段 的扩增并且能够有针对性的提高短片段的扩增效率,降低了核酸片段化及核酸片段化的影 响;所述无dU容忍性的DNA聚合酶,该DNA聚合酶对于dU碱基不耐受,不能识别DNA模板上的U 碱基,因此受细胞分选的固定剂作用C-U的错误突变在扩增阶段就会被排除,极大的降低了 扩增错误率。 在一些实施方案中,所述优化的扩增缓冲液进一步包括:45-55mM的Tris-HCl (pH7.4)、23-27mM的KCl、18-22mM的MgCl2、3-7mM的(NH4)2SO4、0.8-1.2mM的dNTPs、22-27μM 的Pre-primer,所述优化的引物浓度为:9-18ng/ul的泓迅181114引物。 在一些实施方案中,所述无dU容忍性的DNA聚合酶为:0.05-0.1U/uL的Deep Vent EXO- R DNA polymerase。 在一些实施方式中,所述试剂盒用于单细胞全基因组扩增、和/或构建单细胞全基 因组DNA文库。 在一些实施方案中,所述扩增产物可用于高通量测序平台测序、芯片平台测序及 数据分析。 附图说明 结合以下附图一起阅读时,将会更加充分地描述本申请内容的上述和其他特征, 本申请内容将会得到更加明确和详细的说明。可以理解,这些附图仅描述了本申请内容的 若干实施方式,因此不应认为是对本申请内容范围的限定。 图1显示了本发明的一个技术方案的流程图。 图2显示了ChromInst单细胞全基因组扩增的原理图。 图3显示了MDA扩增与本发明方法扩增的碱基错误频率。 5 CN 111575346 A 说 明 书 4/10 页 图4显示了ChromInst试剂盒与本发明方法扩增的CNV分析结果。 发明详述 在详细描述本发明之前,应了解,本发明不受限于本说明书中的特定方法及实验 条件,因为所述方法以及条件是可以改变的。另外,本文所用术语仅是供说明特定实施方案 之用,而不意欲为限制性的。 本发明提供一种针对细胞分选后的单细胞全基因组扩增方法,以及提供一种针对 细胞分选后的单细胞全基因组扩增的试剂盒。 在本发明之前,对目标微量细胞的细胞分选,会造成细胞损伤、核酸片段化、DNA- 蛋白质交联以及碱基变异,从而影响扩增效率、扩增均匀性以及导致扩增错误率较高。本申 请人发明人通过温和的裂解体系和优化的扩增体系,降低细胞分选的干扰,获得更高的扩 增效率、更好的扩增均匀性、且扩增错误率低。温和裂解体系采用温和裂解配方,同时降低 裂解温度并且延长裂解时间,减少细胞损伤和DNA-蛋白质交联;优化的扩增体系为优化的 ChromInst扩增体系具有优化的扩增缓冲液和引物浓度、以及无dU容忍性的DNA聚合酶,降 低核酸片段化和扩增错误率。在一些实施方案中,可实现在单细胞水平有效扩增2-4ug的全 基因组产物、覆盖度>90%、重复单一位点扩增成功率>95%。 本申请的一方面,提供一种针对细胞分选后的单细胞全基因组扩增方法,所述方 法包括步骤: (a)对含有目标微量细胞的样本进行细胞分选,获得目标微量细胞;(b)将目标微 量细胞采用温和裂解体系进行裂解,获得DNA,所述温和裂解体系包含温和裂解配方、较低 的裂解温度和较长的裂解时间;(c)对步骤(b)获取的DNA采用优化的ChromInst扩增体系进 行扩增,获得扩增产物,所述优化的ChromInst体系包含优化的扩增缓冲液和引物浓度、以 及无dU容忍性的DNA聚合酶。本申请提供的扩增方法的一种实施方式的图示请见图1。 (a):对含有目标微量细胞的样本进行细胞分选,获得目标微量细胞; 所述含有目标微量细胞的样本为来源于生物样本,进一步的,来源于人类、哺乳动 物,例如生物组织或含有细胞或游离DNA的体液;可以通过已知的方法获取,例如通过口腔 粘膜样本、鼻腔样本、头发、漱口水、脐带血、血液、羊水、体外培养7天内胚胎的无胚胎细胞 的培养液或腔液、内皮细胞等获取。 所述目标微量细胞为人体内含量极低但具有重要研究价值的细胞,例如孕妇外周 血中的胎儿有核红细胞、循环肿瘤细胞、卵细胞、生殖细胞、肿瘤组织细胞等。 所述细胞分选也是本领域公知的,为常规的荧光标记细胞分选技术,例如荧光激 活细胞分选技术(FACS,fluorescence-activated cell sorting)、荧光流式细胞分选技 术。 (b):将目标微量细胞采用温和裂解体系进行裂解,获得DNA; 所述温和裂解体系包含温和裂解配方,减少细胞损伤;同时具有较低的裂解温度 并且较长的裂解时间,减少DNA-蛋白质交联。 在一些实施方案中,所述温和的裂解配方包括表面活性剂和裂解酶。表面活性剂 可以分为离子型、两性和非离子型表面活性剂,一般情况下,两性和非离子型表面活性剂的 裂解效能弱于离子型表面活性剂。示例性的表面活性剂包括:NP-40、吐温、SDS、GHAPS、 TritonX-100、TritonX-114、EDTA、脱氧胆酸钠、胆酸钠、异硫氰酸胍中的一种或多种。示例 6 CN 111575346 A 说 明 书 5/10 页 性的裂解酶可以是蛋白酶K、胃蛋白酶、木瓜蛋白酶等,或其任意组合。 在一些实施方案中,所述温和的裂解配方进一步包括:0.4%-0 .6%的NP-40、 0.2%-0.3%的脱氧胆酸盐、和低浓度的蛋白酶K,进一步的,所述蛋白酶K的浓度由常规的 100μg/mL降低至15-25μg/mL。 在一些实施方案中,所述温和裂解体系的裂解温度较低,进一步的,所述裂解温度 由常规的55℃降低至3-6℃;所述温和裂解体系的裂解时间较长,进一步的,所述裂解时间 由常规的1小时延长至2.5-3.5小时。 (c):对步骤(b)获取的DNA采用优化的ChromInst扩增体系进行扩增,获得扩增产 物。 本发明除了采用温和裂解体系,进一步在ChromInst扩增技术的基础上进行优化, 进一步降低细胞分选对扩增所带来的干扰。所述优化的ChromInst扩增体系包含优化的扩 增缓冲液和引物浓度、以及无dU容忍性的DNA聚合酶。 ChromInst扩增的扩增步骤为本领域公知的,将裂解后获取的基因组DNA加入到反 应混合物中进行预扩增,然后将预扩增产物进行指数扩增,获得扩增产物。ChromInst单细 胞全基因组扩增的原理见图2。 本发明进一步在ChromInst扩增技术的基础上进行优化,ChromInst扩增的主体扩 增步骤不变,但是进一步优化了预扩增和指数扩增中的扩增缓冲液和引物浓度、以及使用 无dU容忍性的DNA聚合酶,所述优化的扩增缓冲液和引物浓度,更适合核酸短片段的扩增并 且能够有针对性的提高短片段的扩增效率,降低了核酸片段化及核酸片段化的影响;所述 无dU容忍性的DNA聚合酶,该DNA聚合酶对于dU碱基不耐受,不能识别DNA模板上的U碱基,因 此受细胞分选的固定剂作用C-U的错误突变在扩增阶段就会被排除,极大的降低了扩增错 误率。 在一些实施方案中,所述优化的扩增缓冲液进一步包括:45-55mM的Tris-HCl (pH7.4)、23-27mM的KCl、18-22mM的MgCl2、3-7mM的(NH4)2SO4、0.8-1.2mM的dNTPs、22-27μM 的Pre-primer,所述优化的引物配比为:9-18ng/ul的泓迅181114引物。 在一些实施方案中,所述无dU容忍性的DNA聚合酶为:0.05-0.1U/uL的Deep Vent EXO- R DNA polymerase,可以有效的避免由于脱氨基作用导致的G/C到T/A的假阳性突变。 在一些实施方案中,所述扩增产物可用于高通量测序平台测序、芯片平台测序及 数据分析。 所述测序方法为本领域公知的,例如杂交测序法(SBH)、连接酶测序法(SBL)、定量 增量荧光核酸增加测序法(QIFNAS)、逐步连接和切割法、分子信标法、焦磷酸测序法、多重 测序法、聚合群体(POLONY)测序法、摆动测序法、TaqMan报告分子探针消化法、和等位基因 特异的寡核苷酸连接分析法等。例如,高通量测序的方法,高通量测序的方法通过在一次测 序反应中平行地对几万个、几十万个、几百万个、几千万个、甚至上亿个这样的短片段测序, 可以大大提高测序的通量、缩短测序所需的时间。将测得的短片段的序列通过软件进行数 据处理,可以拼接成完整的序列。本领域已知多种高通量测序平台,例如Roche 454、 Illumina Solexa、AB-SOLiD、Helicos、Polonator平台技术等。 所述数据分析也是本领域公知的,例如单核苷酸多态性(SNP)分析、短串联重复序 列(STR)分析、限制性片段长度多态性(RFLP)分析、可变数目串联重复序列(VNTRs)分析、复 7 CN 111575346 A 说 明 书 6/10 页 杂重复序列(CTR)分析或微卫星分析等。 在一些实施方式中,本申请的方法得到的扩增产物还可以用于医学分析和/或诊 断分析。例如,可以对个体的生物样品用本申请的方法进行扩增,分析扩增产物中在感兴趣 的基因或DNA序列中是否存在突变、缺失、插入或染色体之间的融合等异常情况,从而评估 该个体患上某种疾病的风险、疾病的进展阶段、疾病的基因分型、疾病的严重程度、或者该 个体对某种疗法反应的可能性。可以使用本领域已知的适当的方法对感兴趣的基因或DNA 序列进行分析,例如但不限于,通过核酸探针杂交、引物特异性扩增、对感兴趣的序列测序、 单链构象多态性(SSCP)等。 本申请的另一方面,提供一种针对细胞分选后的单细胞全基因组扩增的试剂盒, 所述试剂盒包括:温和的裂解配方、优化的ChromInst扩增体系。 在一些实施方案中,所述温和的裂解配方进一步包括:0.4%-0 .6%的NP-40、 0.2%-0.3%的脱氧胆酸盐、和低浓度的蛋白酶K,进一步的,蛋白酶K的浓度由常规的100μ g/mL降低至15-25μg/mL;目标微量细胞进行裂解时,与温和裂解配方相配合的,需要使用较 低的裂解温度和较长的裂解时间,所述裂解温度由常规的55℃降低至3-6℃,所述裂解时间 由常规的1小时延长至2.5-3.5小时。 在一些实施方案中,所述优化的ChromInst扩增体系包含优化的扩增缓冲液和引 物浓度、以及无dU容忍性的DNA聚合酶,所述优化的扩增缓冲液和引物浓度,更适合核酸短 片段的扩增并且能够有针对性的提高短片段的扩增效率,降低了核酸片段化及核酸片段化 的影响;所述无dU容忍性的DNA聚合酶,该DNA聚合酶对于dU碱基不耐受,不能识别DNA模板 上的U碱基,因此受细胞分选的固定剂作用C-U的错误突变在扩增阶段就会被排除,极大的 降低了扩增错误率。 在一些实施方案中,所述优化的扩增缓冲液进一步包括:45-55mM的Tris-HCl (pH7.4)、23-27mM的KCl、18-22mM的MgCl2、3-7mM的(NH4)2SO4、0.8-1.2mM的dNTPs、22-27μM 的Pre-primer,所述优化的引物配比为:9-18ng/ul的泓迅181114引物。 在一些实施方案中,所述无dU容忍性的DNA聚合酶为:0.05-0.1U/uL的Deep Vent EXO- R DNA polymerase。 在一些实施方式中,所述试剂盒用于单细胞全基因组扩增、和/或构建单细胞全基 因组DNA文库。 在一些实施方案中,扩增产物可用于高通量测序平台测序、芯片平台测序及数据 分析。 “和/或”在本申请中,用于连接两个或多个可选项时,应理解为意指可选项中的任 一项或可选项中的任意两项或多项。 “包含”或“包括”在本申请中,意指包括所述的要素、整数或步骤,但是不排除任意 其他要素、整数或步骤。在本文中,当使用“包含”或“包括”时,除非另有指明,否则也涵盖由 所述及的要素、整数或步骤组成的情形。 “覆盖率”是指,基因组或染色体区段上获知序列信息的序列部分占整个组或区段 的比例。在一些实施方案中,覆盖度是指,例如通过测序测到序列信息的碱基数占所检测区 域的总碱基数的比例。例如,在测序检测全基因组序列时,由于存在大片段拼接的缺口 (gap)、测序读长有限、重复序列等问题,测序后得到的基因组序列通常无法完全覆盖基因 8 CN 111575346 A 说 明 书 7/10 页 组的所有区域,此时,覆盖度就是最终得到的测序碱基数占整个基因组碱基数的比例。在另 一些实施方案中,覆盖度是指,就所检测的区域而言,(例如通过SNP芯片或测序分析)测到 序列信息的基因位点(例如SNP位点或基因变异位点)的数目,占该区域中所检测的总基因 位点数的比例。所检测区域可以是全基因组、特定染色体、或特定染色体区段、或转录物组、 或特定转录区域。 “Read”也称为“读长”,测序数据中每一条序列就是一个read。 “芯片”,是指利用所述芯片杂交后获得的信号(通常为荧光信号)能够判断某个位 点的基因型。在实际的研究中,SNP芯片会因芯片厂家、型号等的不同而包含不同的SNP位 点。例如Affymetrix公司和Illumina公司生产的人类芯片包含不同的SNP集。 实施例 描述以下实施例以辅助对本发明的理解。不意在且不应当以任何方式将实施例解 释成为限制本发明的保护范围。 实施例1:高通量测序平台进行单细胞全基因组测序 步骤(a):对含有目标微量细胞的样本进行细胞分选,获得目标微量细胞。 取人体肿瘤组织样本,经过荧光激活细胞分选技术(FACS)分选捕获目标微量细 胞,获得目标微量细胞为CD133 肿瘤干细胞。 步骤(b):对步骤(a)获得的目标微量细胞采用温和裂解体系进行裂解,获得DNA。 获得的CD133 肿瘤干细胞,对照组采用常规的裂解液进行裂解,实验组采用本发 明记载的裂解方法进行裂解。对照组采用Qiagen REPLI-g Single Cell kit试剂盒进行裂 解,获得DNA,操作步骤参见产品说明书。实验组将获取的目标微量细胞加入到温和裂解配 方中,该温和裂解配方包括:0.5%NP-40、0.25%脱氧胆酸盐、和20μg/mL的蛋白酶K,同时, 裂解温度为4℃,裂解时间为3小时,获得DNA。 步骤(c):对步骤(b)获取的DNA采用优化的ChromInst扩增体系进行扩增,获得扩 增产物。 获得的DNA,对照组采用商品化的MDA试剂盒:Qiagen REPLI-g Single Cell kit 试剂盒,进行单细胞全基因组扩增,实验组采用本发明记载的扩增方法进行单细胞全基因 组扩增。 对照组采用Qiagen REPLI-g Single Cell kit试剂盒进行单细胞全基因组扩增, 获得扩增产物,操作步骤参见产品说明书。实验组将获取的DNA加入到反应混合液中进行预 扩增,反应混合液包括:50mM的Tris-HCl(pH7 .4)、25mM的KCl、20mM的MgCl2、5mM的(NH4) 2SO4、1.0mM的dNTPs、25μM的Pre-primer、12ng/ul的泓迅181114引物、0.08U/uL的Deep Vent EXO- R DNA polymerase,预扩增程序为: 9 CN 111575346 A 说 明 书 8/10 页 预扩增程序后,将预扩增产物进行指数扩增得到扩增产物,指数扩增程序为: 步骤(d):高通量测序及数据分析。 对照组及实验组获得的扩增产物,采用商品化NGS建库试剂盒:NEBNext Ultra II DNA试剂盒构建高通量测序文库,并在Illumina Novaseq测序平台上进行全基因组测序;测 序数据获取后,采用FastQC软件过滤低质量的reads,用bwa软件将高质量的reads与参考基 因组做比对,最后用GATK软件进行SNP识别(SNP calling),其中,FastQC软件、bwa软件以及 GATK软件均为商品化的软件,具体为开源软件。 将对照组采用MDA扩增和实验组采用本发明方法扩增,然后均测序得到的SNP进行 比较,比较两种扩增方法导致的碱基错误频率,结果见附图图3,从图3中可以看出,采用本 发明的单细胞全基因组扩增方法比MDA扩增比较,本发明扩增方法的碱基错误频率明显低 于MDA扩增。 实施例2:芯片平台进行单细胞全基因组测序 步骤(a):对含有目标微量细胞的样本进行细胞分选,获得目标微量细胞。 取孕妇外周血,经过荧光激活细胞分选技术(FACS)分选捕获目标微量细胞,获得 目标微量细胞为胎儿有核红细胞。 步骤(b):对步骤(a)获得的目标微量细胞采用温和裂解体系进行裂解,获得DNA。 10 CN 111575346 A 说 明 书 9/10 页 获得的胎儿有核红细胞,对照组采用常规的裂解液进行裂解,实验组采用本发明 记载的裂解方法进行裂解。对照组采用Qiagen REPLI-g Single Cell kit试剂盒进行裂 解,获得DNA,操作步骤参见产品说明书。实验组将获取的目标微量细胞加入到温和裂解配 方中,该温和裂解配方包括:0.5%NP-40、0.25%脱氧胆酸盐、和20μg/mL的蛋白酶K,同时, 裂解温度为4℃,裂解时间为3小时。 步骤(c):对步骤(b)获取的DNA采用优化的ChromInst扩增体系进行扩增,获得扩 增产物。 获得的DNA,对照组采用商品化的ChromInst试剂盒进行单细胞全基因组扩增,实 验组采用本发明记载的扩增方法进行单细胞全基因组扩增。 对照组ChromInstTM基因测序通用文库试剂盒(亿康基因)进行单细胞全基因组扩 增,获得扩增产物,操作步骤参见产品说明书。实验组将获取的DNA加入到反应混合液中进 行预扩增,反应混合液包括:50mM的Tris-HCl(pH7 .4)、25mM的KCl、20mM的MgCl2、5mM的 (NH4)2SO4、1.0mM的dNTPs、25μM的Pre-primer、16ng/ul的泓迅181114引物、0.08U/uL的Deep Vent EXO- R DNA polymerase,预扩增程序为: 预扩增程序后,将预扩增产物进行指数扩增得到扩增产物,指数扩增程序为: 步骤(d):高通量测序及数据分析。 对照组及实验组获得的扩增产物,使用Illumina公司的Infinium Asian Screening Array芯片对扩增产物进行检测;芯片原始数据获取后,使用Illumina公司的 GenomeStudio软件中的基因分型功能模块进行数据分析。 11 CN 111575346 A 说 明 书 10/10 页 比较ChromInst试剂盒与本发明方法扩增产物的CNV分析结果,结果见附图图4,图 4左图为ChromInst试剂盒扩增产物的CNV分析结果,右图为本发明方法扩增产物的CNV分析 结果,对比可见,本发明方法的扩增的均一性更好。 尽管本发明已公开了多个方面和实施方式,但是其它方面和实施方式对本领域技 术人员而言将是显而易见的。本发明公开的多个方面和实施方式仅用于举例说明,其并非 旨在限制本发明,本发明的实际保护范围以权利要求为准。 12 CN 111575346 A 说 明 书 附 图 1/3 页 图1 图2 13 CN 111575346 A 说 明 书 附 图 2/3 页 图3 14 CN 111575346 A 说 明 书 附 图 3/3 页 图4 15