技术摘要:
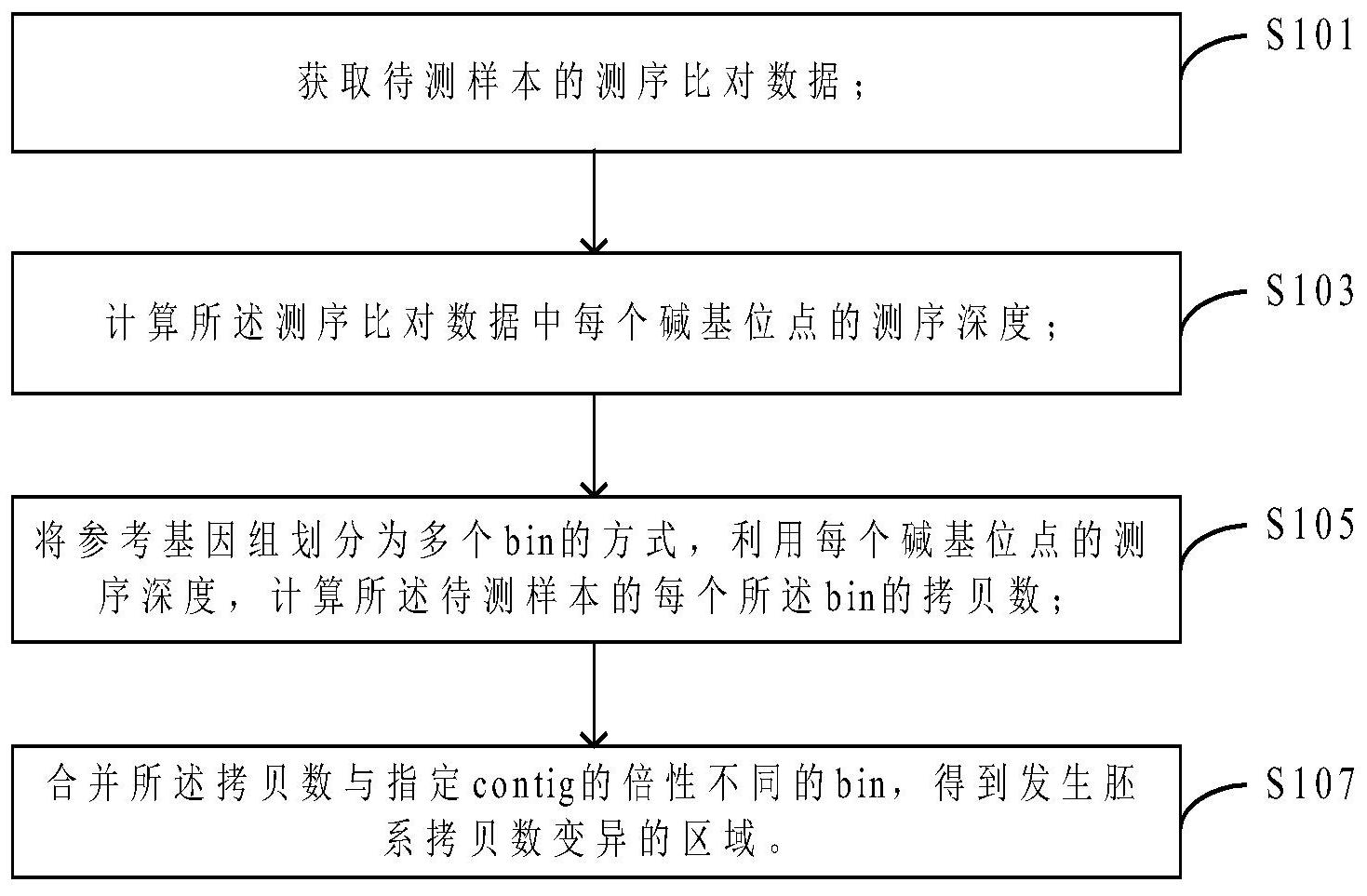
本发明提供了一种拷贝数变异的检测方法和装置。该检测方法包括:获取待测样本的测序比对数据;计算测序比对数据中每个碱基位点的测序深度;将参考基因组划分为多个bin的方式,利用每个碱基位点的测序深度,计算待测样本的每个bin的拷贝数;合并拷贝数与指定contig的倍 全部
背景技术:
CNV是指长度大于1kb的拷贝数多态,是基因组结构变异(SV)的一种,包括拷贝数 的缺失(deletion)、插入(insertion)、重复(duplication)和复杂多位点变异(complex muti-site variants)。CNV的产生机制之一是DNA重组,包括非等位同源重组(Nonallelic homologous recombination,NAHR)和非同源末端连接(Nohomologous end-joining,NHEJ) 等。DNA重组引起的CNV可以从以下几个方面影响基因的表达:(1)基因剂量;(2)基因断裂; (3)基因融合;(4)位置效应;(5)隐型等位基因显性化等。 CNV的检测有目前常用的有以下几种方法: 多重连接扩增技术(multiplex ligation dependent probe amplification, MLPA),针对每个待测靶基因设计相邻的两个探针,探针通过通用引物与靶序列配对杂交 后,两个相邻的探针通过连接反应相连,连接产物的量与靶基因的拷贝数成正比。连接产物 经PCR扩增后可以根据电泳结果分析基因的拷贝数。 芯片技术,这种技术是将感兴趣的靶点做成微阵列芯片,对基因组中关键区域进 行系统性的扫描。目前应用较为广泛的芯片主要有比较基因组杂交芯片(comparative genomic hybridization,CGH)和SNP芯片。这种技术只能检测已知的CNV。
技术实现要素:
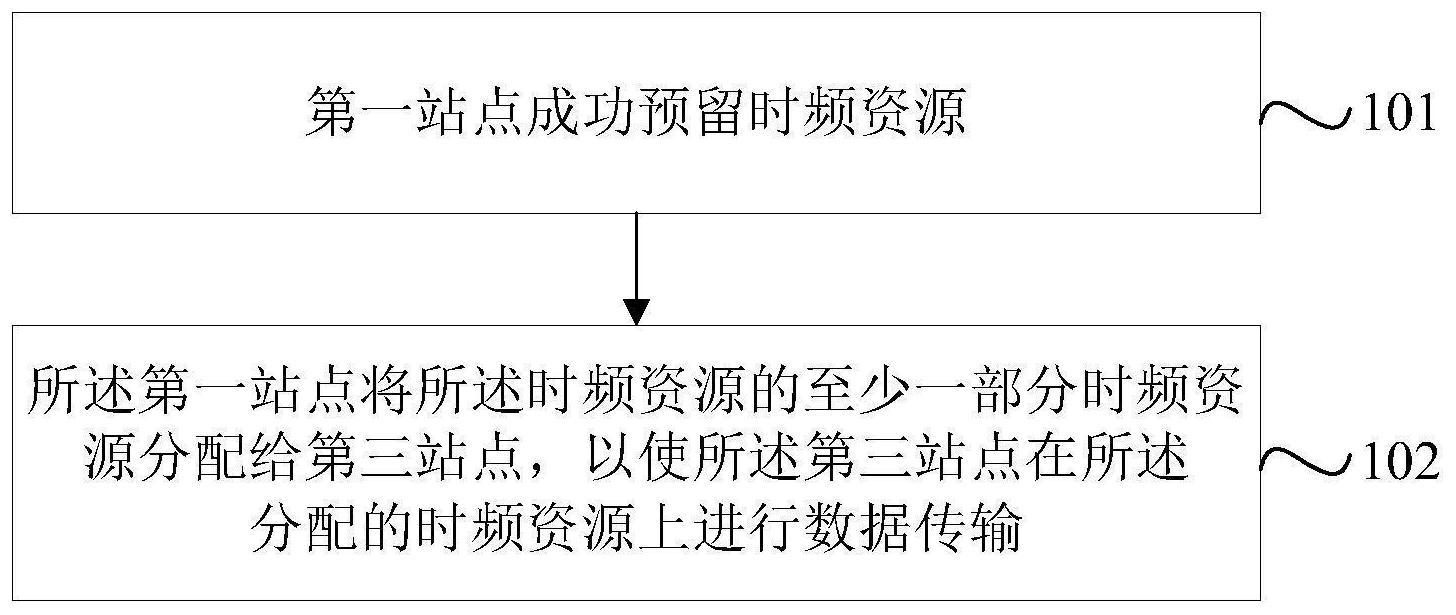
本发明的主要目的在于提供一种拷贝数变异的检测方法和装置,以解决现有技术 中对突变检测的灵敏度低的问题。 为了实现上述目的,根据本发明的一个方面,提供了一种拷贝数变异的检测方法, 该检测方法包括:获取待测样本的测序比对数据;计算测序比对数据中每个碱基位点的测 序深度;将参考基因组划分为多个bin的方式,利用每个碱基位点的测序深度,计算待测样 本的每个bin的拷贝数;合并拷贝数与指定contig的倍性不同的bin,得到发生胚系拷贝数 变异的区域。 进一步地,获取待测样本的测序比对数据包括:获取待测样本的测序原始数据;对 测序原始数据进行质控,得到测序比对数据;优选地,对测序原始数据进行质控,得到测序 比对数据包括:对测序原始数据进行预处理,去除如下至少一种reads:(1)含有接头的 reads;(2)质量低于阈值的reads,得到预处理数据;将预处理数据与参考基因组序列比对, 得到比对结果数据;对比对结果数据进行过滤处理,过滤去除具有重复比对结果的reads, 得到测序比对数据;更优选地,对比对结果数据进行过滤处理,进一步包括过滤去除目标捕 获区域外的reads。 进一步地,在将参考基因组划分为多个bin的方式,计算待测样本的每个bin的拷 4 CN 111599407 A 说 明 书 2/7 页 贝数之前,检测方法还包括,将参考基因组划分为多个bin,并对待测样本的每个bin的测序 深度进行归一化处理;然后利用归一化之后的测序深度计算每个bin的拷贝数;优选地,归 一化处理包括:根据用于构建基线的样本在每个bin的测序深度,利用主成分分析法建立归 一化模型;利用归一化模型对待测样本中的每个bin的测序深度进行归一化;优选地,利用 归一化之后的测序深度,采用Viterbi算法计算待测样本的每个bin的拷贝数。 进一步地,合并拷贝数与指定contig的倍性不同的bin,得到发生拷贝数变异的区 域包括:根据每个bin的拷贝数,筛选拷贝数与指定contig的倍性不同的bin,得到差异bin 集;将差异bin集中属于同一基因同一外显子的多个不同的bin进行合并,得到发生拷贝数 变异的区域。 根据本申请的第二个方面,提供了一种拷贝数变异的检测装置,该检测装置包括: 获取模块,用于获取待测样本的测序比对数据;深度计算模块,用于计算测序比对数据中每 个碱基位点的测序深度;拷贝数计算模块,用于将参考基因组划分为多个bin的方式,利用 每个碱基位点的测序深度,计算待测样本的每个bin的拷贝数;合并模块,用于合并拷贝数 与指定contig的倍性不同的bin,得到发生胚系拷贝数变异的区域。 进一步地,获取模块包括:获取子模块,用于获取待测样本的测序原始数据;质控 模块,用于对测序原始数据进行质控,得到测序比对数据;优选地,质控模块包括:去除模 块,用于对测序原始数据进行预处理,去除如下至少一种reads:(1)含有接头的reads;(2) 质量低于阈值的reads,得到预处理数据;比对模块,用于将预处理数据与参考基因组序列 比对,得到比对结果数据;第一过滤模块,用于对比对结果数据进行过滤处理,过滤去除具 有重复比对结果的reads,得到测序比对数据;更优选地,质控装置还包括第二过滤模块,用 于对比对结果数据进行过滤处理,以过滤去除目标捕获区域外的reads。 进一步地,拷贝数计算模块包括:归一化模块,用于将参考基因组划分为多个bin, 并对待测样本的每个bin的测序深度进行归一化处理;拷贝数计算子模块,用于利用归一化 之后的测序深度计算每个bin的拷贝数;优选地,归一化模块包括:模型建立模块,用于根据 用于构建基线的样本在每个bin的测序深度,利用主成分分析法建立归一化模型;归一化子 模块,用于利用归一化模型对待测样本中的每个bin的测序深度进行归一化;更优选地,拷 贝数计算子模块为Viterbi模块。 进一步地,合并模块包括:筛选模块,用于根据每个bin的拷贝数,筛选拷贝数与指 定contig的倍性不同的bin,得到差异bin集;合并子模块,用于将差异bin集中属于同一基 因同一外显子的多个不同的bin进行合并,得到发生拷贝数变异的区域。 根据本申请的第三个方面,提供了一种存储介质,该存储介质包括存储的程序,其 中,在程序运行时控制存储介质所在设备执行上述任一种拷贝数变异的检测方法。 根据本申请的第四个方面,提供了一种处理器,该处理器用于运行程序,其中,程 序运行时执行上述任一种拷贝数变异的检测方法。 应用本发明的技术方案,通过采用基于以bin的方式获得每个bin的倍性(即拷贝 数),然后通过与指定的contig的倍性进行比较,对被划分为多个不同的bin,但属于同一基 因相同染色体的差异bin合并,从而检测出长度超过1000bp的基因外显子的缺失或重复。与 现有技术中的芯片方法相比,该方法检测CNV具有更高的覆盖度、分辨率及更准确的拷贝数 评估,不仅能够检测某些已知位点的拷贝数变异情况,而且可以检测未知的拷贝数变异情 5 CN 111599407 A 说 明 书 3/7 页 况,提高检测的灵敏度。 附图说明 构成本申请的一部分的说明书附图用来提供对本发明的进一步理解,本发明的示 意性实施例及其说明用于解释本发明,并不构成对本发明的不当限定。在附图中: 图1示出了根据本发明的一种优选的实施例的拷贝数变异的检测方法的流程示意 图; 图2示出了根据本发明的实施例2的拷贝数变异的检测方法的详细流程示意图; 图3示出了根据本发明的实施例3对已知样本的拷贝数变异的检测结果验证图; 图4示出了根据本发明的一种优选的实施例的拷贝数变异的检测装置的结构示意 图。