技术摘要:
本发明公开一种基于深度学习的生物医学实体识别方法,包括步骤:使用词嵌入将输入序列向量化表示,以及通过CNN模型获取字符特征向量;将词嵌入向量以及通过CNN模型所获取的字符特征向量作为HDL‑ATT模型的输入向量;利用双向长短时记忆模型BLSTM分别从序列的两侧同时对 全部
背景技术:
随着生物技术的迅速发展和人类健康医疗不断重视,生物医学领域的研究得以迅 速发展,相关文献也呈井喷式地快速增长,依据文献了解前人的研究进展和进行必要的学 术交流是提升研究水平,推进研究进展的必需途径,仅依据专业研究人员个体的能力是难 以从海量的文献中进行学习的,因此发展生物医学文本挖掘显得十分必要,而相关的文本 实体识别是生物医学文本挖掘技术的基石和重要的一步。 深度学习以其强大的计算能力,自动特征表示能力和类似人脑结构的学习能力迅 速发展。神经网络可以更好地挖掘文本的语义信息,基于神经网络的深度学习关系提取方 法可以自动学习实体关系的有效特征,并且无需手动定义特征模板,因此,基于深度学习的 实体识别方法在命名实体识别任务中较传统方法往往具有更好的性能。 生物医学实体名识别与实体相互作用关系抽取两个任务是最近几年的研究热点, 相关的研究已经取得了一定进展,但也存在一些问题。
技术实现要素:
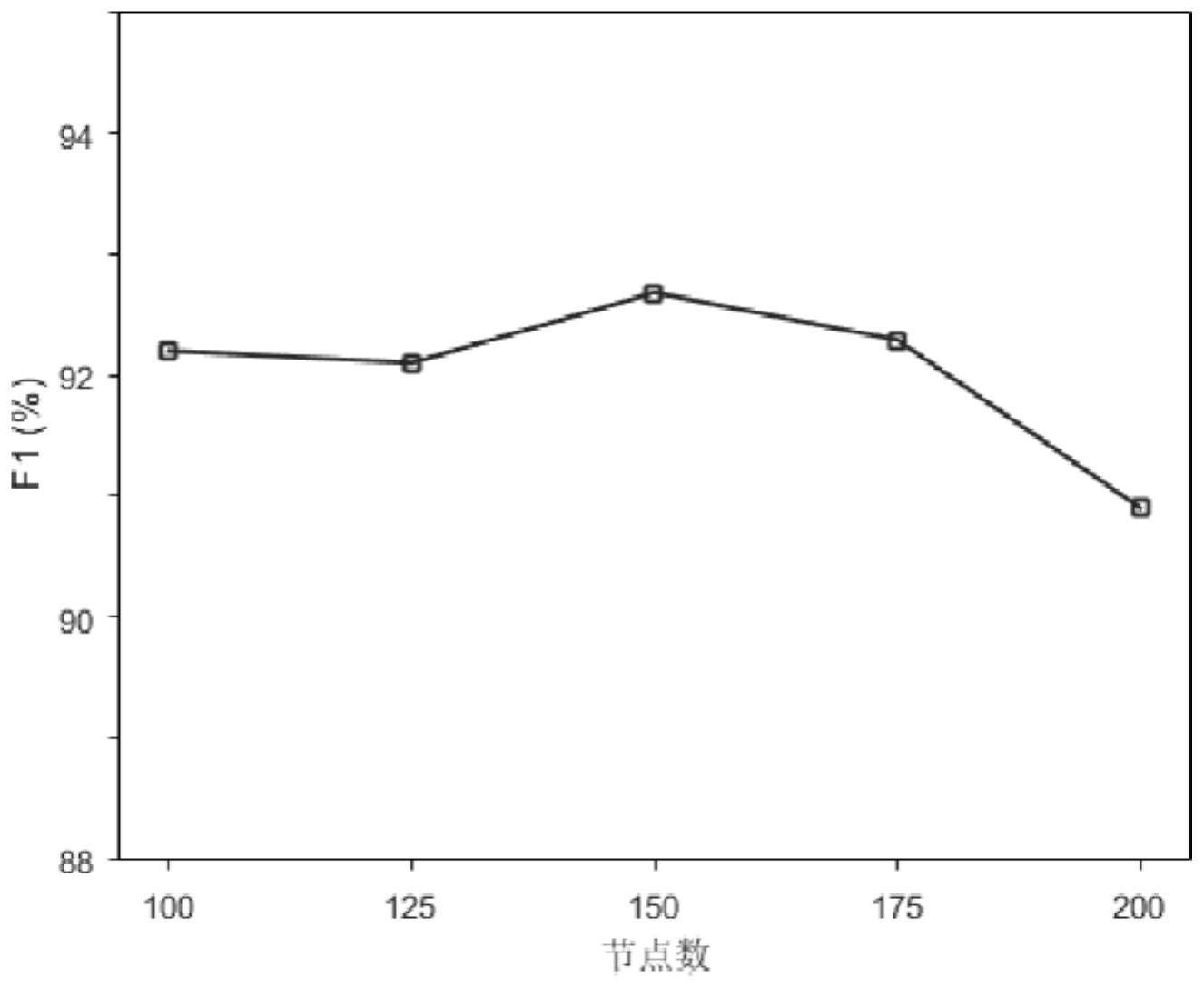
本发明的目的是针对现有技术中存在的技术缺陷,而提供一种基于深度学习的生 物医学实体识别方法。 为实现本发明的目的所采用的技术方案是: 一种基于深度学习的生物医学实体识别方法,包括步骤: 使用词嵌入将输入序列向量化表示,以及通过CNN模型获取字符特征向量; 将词嵌入向量以及通过CNN模型所获取的字符特征向量作为HDL-ATT模型的输入 向量; 利用双向长短时记忆模型BLSTM分别从序列的两侧同时对序列进行处理,对上下 文信息建模,捕捉输入向量实体关系的文本序列中的句子级别的特征;同时利用自注意力 机制层引入篇章信息,捕捉与目标领域的实体识别有密切联系的文本片段信息; 将通过自注意力机制层所得到的文本表示作为CRF层的输入,CRF层根据自注意力 层的输入特征为序列的实体进行分类,得到最终的序列输出。 其中,所述自注意力机制层的表示向量由基于上下文的词语表示和基于上下文的 词性表示组成。 其中,所述通过CNN模型获取字符特征向量的方法如下:首先收集字符集合,字符 集采用随机排序方法行排列,构成基于字符的查找表,根据查找表CNN模型获得基于字符向 量的特征向量表示。 本发明结合应用BLSTM网络和CRF模型,同时加入ATT自注意力机制构建了一个 3 CN 111581974 A 说 明 书 2/9 页 HDL-ATT混合模型。这个模型不需要手动地建立复杂的特征,应用词向量和基于字符的特征 向量作为输入向量,经过BLSTM对输入序列向量进处理,输出的向量值利用ATT进行加权赋 值再加载到CRF模型中,得到基于实体识别的分类结果。 利用DDIExtraction 2011和DDIExtraction 2013评价模型,验表明这个模型在药 名识别上,其性能和其他研究模型的性能具有一定的优势。 模型应用在EPI语料集和JNLPBA 2012语料上,发明表明在蛋白质等生物医学实体 的识别上,其性能比当前最佳的性能有显著提升,也说明该模型在生物文本识别领域的鲁 棒性和优越性。 附图说明 图1是为不同隐藏层节点数对HDL-ATT模型结果的影响图; 图2是不同词向量初始化策略对HDL-ATT模型结果的影响图; 图3是本发明所用的识别网络模型的结构原理图。