技术摘要:
本发明公开了一种结合网络聚类方法的全局多网络比对方法。本发明一种结合网络聚类方法的全局多网络比对方法,包括:步骤1.读取蛋白质相互作用网络数据和不同物种间的序列相似性数据,以及设定参数α和β,构建蛋白质相互作用网络G和序列相似性网络S;步骤2.对序列相似 全部
背景技术:
21世纪以来,不同研究领域,如社交网络、商业交易和分子生物学等,可获得的数 据量都出现了爆炸式增长。将蛋白质及其相互作用以网络(图)的形式表示并研究新的策略 对其进行分析,是目前的一个研究热点。在蛋白质相互作用网络中,节点表示蛋白质,边表 示两个蛋白质之间的相互作用。 基于蛋白质相互作用网络的比对研究较为广泛,主要分为成对(两个网络)网络比 对和多(三个及以上)网络比对。早期的网络比对算法多为成对比对,旨在寻找两个网络中 相似度最高的映射节点。自2008年起网络比对的研究逐渐转移到多网络比对算法上,多网 络比对算法可以同时得到多个网络间节点的映射关系,因此多网络比对能够获得更加深入 的生物意义。 由于网络比对问题可看为子图同构问题,因此网络比对是NP-完全问题,一直以来 网络比对通过采用启发式方法来解决这个问题。早期多使用贪心算法进行比对,经过多年 发展越来越多的方法用于网络比对当中,例如,匈牙利算法、种子与扩展匹配方法、模拟退 火算法、遗传算法等等,近几年也有采用深度学习方法用来解决网络比对问题。 传统技术存在以下技术问题: 1、蛋白质相互作用网络的全局多网络比对研究起源于2009年,由Liao(IsoRankN: spectral methods for global alignment of multiple protein networks[J] .Bioinformatics,25(12):i253-i258.)等人提出的IsoRankN算法。它通过建立不同网络间 节点的相似性得分矩阵,再利用频谱聚类方法生成多网络比对结果。但是作为一个早期的 算法,它与近几年新提出的多网络比对算法相比,在拓扑意义和生物功能意义上大多略逊 一筹。由于这是一个经典的多网络比对算法,后期提出的许多算法还是会以此方法为标准 进行比较。 2、2013年Sahraeian(SMETANA:Accurate and Scalable Algorithm for Probabilistic Alignment of Large-Scale Biological Networks[J].Plos One,8(7): e67995)等人提出的SMETANA算法。首先利用半马尔科夫随机游走模型计算不同网络节点间 的相似得分矩阵,再通过两次概率一致性转移提高节点间的比对概率,最后利用贪心的种 子与扩展方法构建最终比对。许多数据集的实验结果表明SMETANA是一个能够获得较好拓 扑结果的多网络比对算法,但是其获得较好拓扑意义(较高的保守边比例)是以部分节点间 的功能相似性为代价的,也就是说SMETANA大多只能够获得拓扑意义较好而生物功能意义 欠佳的比对结果。 3、2014年Ferhat A(BEAMS:backbone extraction and merge strategy for the global many-to-many alignment of multiple PPI networks,Bioinformatics,2014,30 5 CN 111599406 A 说 明 书 2/9 页 (4) ,531-539.)等人提出的BEAMS算法是一种以种子与扩展为框架的全局多网络比对算法, 通过在网络中搜索加权最大团(backbone)的方式产生比对结果,它主要分为backbone提取 和合并两个部分。BEAMS算法是一个能够获得较好生物功能意义的比对算法,但是比对结果 的拓扑意义(保守边比例不高)却不好,且在比对过程中过渡依赖节点间的序列相似性信 息,因此不能够很好地平衡拓扑和生物功能意义。
技术实现要素:
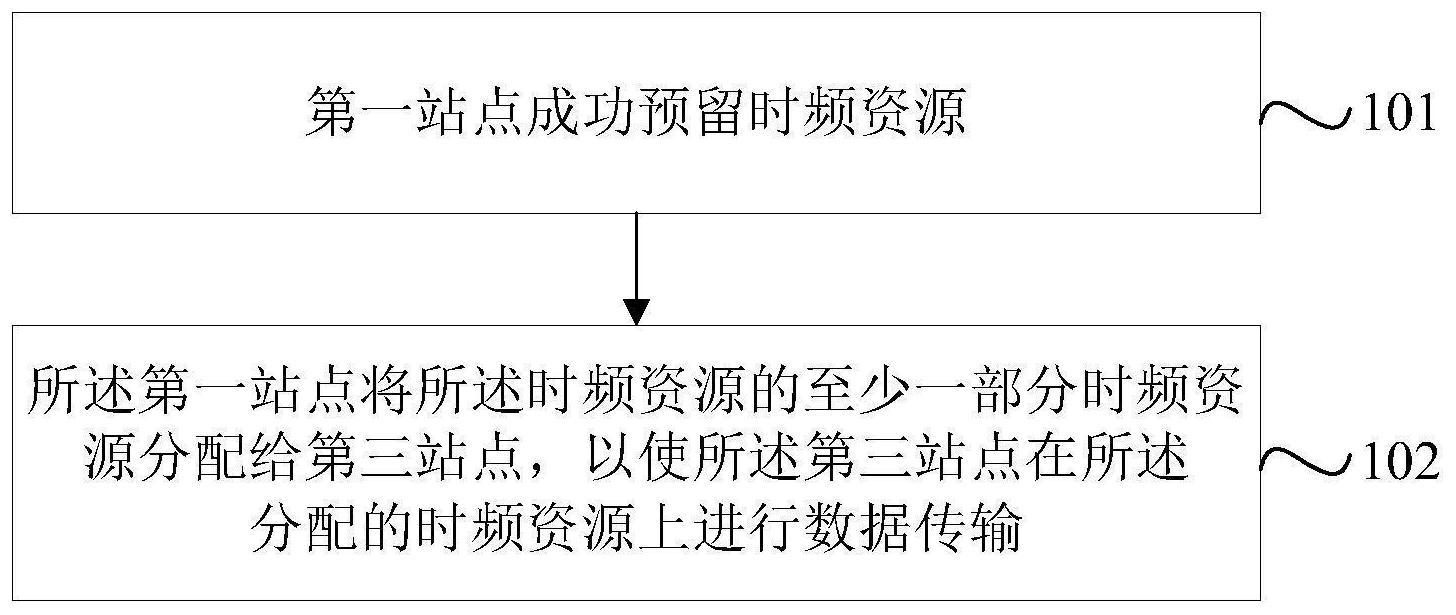
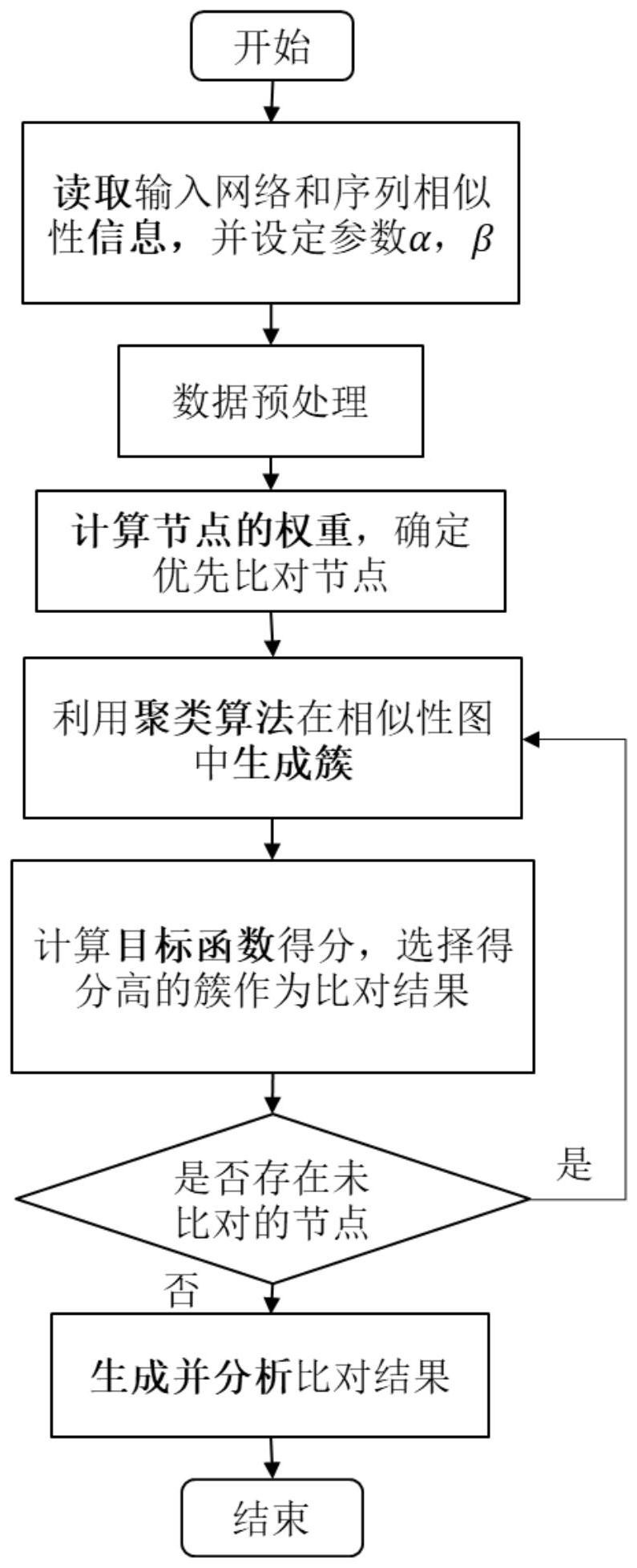
本发明要解决的技术问题是提供一种结合网络聚类方法的全局多网络比对方法, 序列相似性数据的不完整对网络比对准确性的影响;降低在较大数据规模下选择搜索比对 节点的复杂度;基因复制导致的每个网络中有多个相似节点对网络比对准确性的影响;多 网络比对拓扑和生物功能质量的不平衡。 为了解决上述技术问题,本发明提供了一种结合网络聚类方法的全局多网络比对 方法,包括: 步骤1.读取蛋白质相互作用网络数据和不同物种间的序列相似性数据,以及设定 参数α和β,构建蛋白质相互作用网络G和序列相似性网络S; 步骤2.对序列相似性数据进行预处理,利用参数β将序列相似性得分较小的边删 除,得到过滤后的网络Sβ; 步骤3.计算所有网络中每一个节点的权重,根据节点的度和邻居节点,将度小的 节点和边的权重转移到度较大的节点和边上; 步骤4在相应搜索的图中,采用网络聚类算法生成候选簇; 步骤5计算当前候选比对簇的目标函数得分,选择得分最高的候选比对簇作为比 对结果; 步骤6输出比对结果,并对比对结果进行分析。 在其中一个实施例中,步骤1具体如下: 首先,读取用户设定的用来决定拓扑和序列相似性所占比重的参数α和用来过滤 序列相似性信息的参数β;其次确定输入网络个数k,并对蛋白质相互作用网络进行批次读 取,并构建蛋白质相互作用网络G={G1,G2,…,Gk};并读取不同网络间的序列相似性信息; 构建序列信息网络S。 在其中一个实施例中,步骤2具体如下: 根据读取的阈值系数β,对序列相似性信息进行过滤;首先由于序列相似性的信息 量巨大,随着网络数增多数据量呈指数级增长,随之计算难度也会增大;其次由于目前许多 真实物种的序列相似性信息不完整,某些序列信息可能会影响比对结果的准确性;因此在 使用序列相似性辅助比对过程时,需要对序列信息进行过滤;对于序列信息网络S中任一条 边(u,v),若其边的得分小于相应边相关的阈值,则删除网络S中的这条边,最终得到经过过 滤的网络Sβ: w(u,v)<β×max(u,v) (1) 在其中一个实施例中,步骤3具体如下: (a)为网络中的节点和节点间的边设置初始值; 6 CN 111599406 A 说 明 书 3/9 页 (b)将节点度为1的节点的权重转移到它的邻居节点和边上; (c)将节点度大于1且小于10的节点权重转移到它的邻居节点和边上; (d)根据节点的权重和与之相连的边的权重计算节点在网络中的重要性得分,λ表 示计算节点权重时,相关边的权重得分对于节点权重的影响大小; 计算每个节点相关的序列同源得分; (e)计算网络中每一个节点的最终权重得分; Weight(u)=α×importance(u) (1-α)×B(v) (8) (f)结合聚类方法搜索比对: 根据序列相似性信息可以构建一个加权k分完全图S,其中节点表示的是相应网络 中的节点,来自不同物种网络的两节点之间边的权重为序列相似性bitscore值;在相似图S 中通过网络聚类方法,将相似的节点聚集在一个簇中。 在其中一个实施例中,步骤3中,此处为基于种子与扩展方法的聚类方法,分为以 下几个步骤: (a)计算当前搜索网络中节点的加权度,并选择加权度最大的节点作为第一个种 子加入集合S; (b)将第一个种子邻居的权重归一化,并选择权重最高的作为第二个种子加入S; (c)根据前两步生成的种子对在网络中进行扩展,选择与S中节点相接权重和最大 的节点,满足两个约束条件则添加节点,否则结束扩展;当生成了新的候选簇,若簇中包含 的节点来源网络数小于输入网络数,对当前簇进行扩展,提高约束条件,即增大Td,Ts的值; 在其中一个实施例中,步骤5具体如下: 对于每一次迭代中,由上一步骤产生的候选簇,计算该候选簇的目标函数得分,在 候选簇中选择目标函数得分最高的作为此次迭代中产生的比对簇;目标函数公式如下: AS(A)=α×CIQ(A) (1-α)ICQ(A) (11) 7 CN 111599406 A 说 明 书 4/9 页 其中,α是平衡拓扑和序列相似性信息所占比重的参数,通常取值为0.5;CIQ为衡 量簇间拓扑质量的度量,ICQ为衡量簇内节点间的序列相似性的度量: 其中 表示分别在不同的簇Clm,Cln中的节点之间的边的集合,cs(m,n)表示 簇Clm,Cln之间保守边所占比例,计算公式如下: 其中s′m,n表示包含 中的边的网络数,sm,n表示簇Clm,Cln中包含的节点的网 络数;此处,当s′m,n=1时,cs(m,n)=0;否则,由公式13计算。 在其中一个实施例中,其中,下列计算ICQ的公式中,ICQ(Cli)表示仅考虑某一个 簇Cli内节点的得分,ICQ(A)表示比对结果A中考虑到所有簇的得分: 其中,wmax(u)表示与节点u相接的边中权重的最大值,E(Cli)表示在簇Cli中的节点 在Sβ中相接的边的集合。 基于同样的发明构思,本申请还提供一种计算机设备,包括存储器、处理器及存储 在存储器上并可在处理器上运行的计算机程序,所述处理器执行所述程序时实现任一项所 述方法的步骤。 基于同样的发明构思,本申请还提供一种计算机可读存储介质,其上存储有计算 机程序,该程序被处理器执行时实现任一项所述方法的步骤。 基于同样的发明构思,本申请还提供一种处理器,所述处理器用于运行程序,其 中,所述程序运行时执行任一项所述的方法。 本发明的有益效果: 本发明采用的方法能够达到不错的比对效果,且能够产生在拓扑和生物功能意义 上都不错的比对结果。k-coverage为输入网络个数的蛋白质覆盖量表明,本发明采用的聚 类方法能将尽可能多的相似节点比对到同一个簇中,且比对到同一个簇中的节点都具有相 同的生物功能,能够证明本发明的方法的有益效果。 附图说明 图1是本发明结合网络聚类方法的全局多网络比对方法的流程图。 图2是本发明结合网络聚类方法的全局多网络比对方法中的计算节点权重后效果 的一个示例图。 图3是本发明结合网络聚类方法的全局多网络比对方法中的不同算法在合成网络 8 CN 111599406 A 说 明 书 5/9 页 数据集上的实验结果示意图。 图4是本发明结合网络聚类方法的全局多网络比对方法中的合成网络中比对结果 的拓扑和生物指标结果。 图5是本发明结合网络聚类方法的全局多网络比对方法中的真实网络下不同比对 算法的实验结果。 图6是本发明结合网络聚类方法的全局多网络比对方法中的真实网络下不同比对 算法的拓扑和生物指标结果。 图7是本发明结合网络聚类方法的全局多网络比对方法中的不同比对算法的拓扑 和生物指标乘积的对比结果。